
Locked Nucleic Acid Oligonucleotide
Eigenschaften und Anwendung
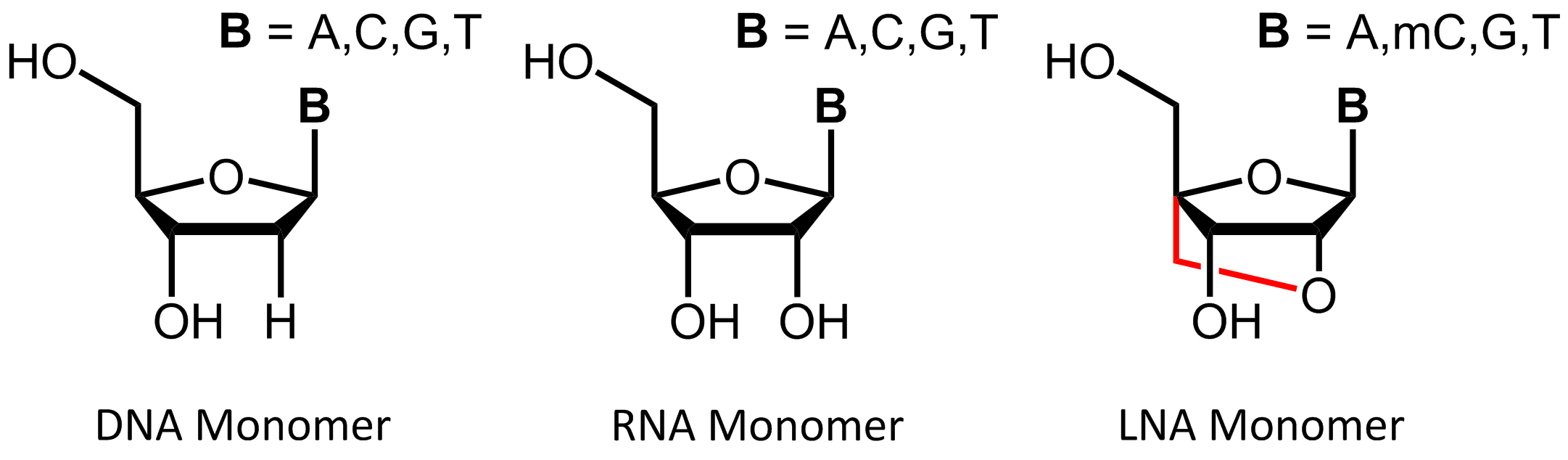
Locked Nucleic Acid (LNA)
Locked Nucleic Acids (LNA) wurden erstmals unabhängig im Jahr 1997 von Jesper Wengel1 und Takeshi Imanishi2 beschrieben und haben sich zu einem festen Bestandteil in Hybridisierungs-basierten Anwendungen entwickelt.
LNA sind eine Klasse von modifizierten RNA-Nucleinsäure Analoga, die über eine zusätzliche Methylenbrücke verfügen. Diese verbindet das Sauerstoffatom am C-2´ der Ribose mit dem 4´-Kohlenstoffatom. Über diese Verknüpfung wird der Ribosering in der 3´-endo Konformation fixiert („locked“) und führt zur charakteristischen
A-RNA Struktur und zur ausschließlichen Bildung von A-Duplexen.
Hybridisierungseigenschaften und Stabilität
Neben den gewöhnlichen Basen Adenin, Guanin und Thymin findet in der Synthese von LNA-Oligonucleotiden auch das Methylcytosin Anwendung, was eine Hybridisierung mit komplementären DNA- oder RNA-Basenpaaren ermöglicht. Die rigide Struktur der LNA, bedingt durch die zusätzliche Methylenbrücke, schränkt die Konformationsfreiheit des LNA-Moleküls derart ein, sodass eine ideale Konformation für die Watson-Crick-Basenpaarung vorliegt. Auf diese Weise kann das LNA-basierte Oligo schneller an einen komplementären Strang binden und auch die Stabilität des resultierenden Doppelstrangs nachweislich erhöhen.3
Eine weitere bemerkenswerte Eigenschaft von LNA ist ihre erhöhte Affinität zu komplementärer DNA und RNA, die LNA-basierte Oligonucleotide ideal für den Nachweis kurzer und sehr ähnlicher DNA- und RNA-Targets macht.
Des Weiteren führt ein Einbau von einzelnen LNA-Basen zu einem Anstieg der Schmelztemperatur.3,6 So zeigten Duplexe aus LNA:DNA-Hybriden in Untersuchungen höhere Annealing-Temperaturen im Vergleich zu entsprechenden DNA:DNA-Duplexen.
Zudem kann der Einbau von LNA die Endo- und Exonucleaseresistenz erhöhen, sodass eine hohe in vivo und in vitro Stabilität der Oligos erzielt werden kann.5 So können beispielsweise drei LNA-Basen an den Enden das Oligonucleotid vor einem nukleolytischen Abbau schützen.7 Setzt sich die Sonde aus alternierenden DNA- und LNA-Bausteinen zusammen (beginnend mit einer LNA-Base am 5´-Ende), zeigt das Konstrukt eine stark erhöhte Stabilität in der Zelle (Nuklease-Resistenz).8
Die Biostabilität LNA-basierter Oligonucleotide in Blut sowie anderen biologischen Flüssigkeiten macht sie für den Einsatz in vivo zu einem interessanten Kandidaten.6
Spezifität und Sensitivität
Die Tatsache, dass LNA-Basen durch das gleiche Phosphatrückgrat verbunden sind wie native DNA oder RNA, macht eine Synthese gemischter Oligonucleotide, bestehend aus LNA/DNA- oder auch LNA/RNA-Basen, möglich. LNA-Oligonucleotide werden unter Verwendung herkömmlicher Phosphoamidit-Chemie synthetisiert, was eine automatisierte Synthese von sowohl reinen LNA- als auch gemischten LNA/DNA- oder LNA/RNA-Oligonucleotiden ermöglicht.4 Je nach Bedarf können in Oligonucleotiden auf diese Weise einzelne oder mehrere LNA-Nucleotide zusammen mit DNA- oder RNA-Nucleotiden eingebaut werden und somit die Spezifität des Oligos verbessern.
Der Einbau eines einzelnen LNA-Nucleotids kann die Schmelztemperatur des Oligos dabei deutlich erhöhen, sodass je nach Anwendung eine präzisere Anpassung der gewünschten Schmelztemperatur von z.B. einem Primer oder einer Sonde möglich ist.
Erhöhung der Schmelztemperatur Tm
Als grober Richtwert erhöht der Einbau von LNA-Nucleotiden in ein DNA-Oligonucleotid (Primer, qPCR Sonde etc.) die Tm um 2-6°C pro substituiertes Nucleotid, in einen RNA-Primer um 3-9,6°C.12 Auf diese Weise erreichen auch kurze LNA-Oligonucleotide eine vergleichsweise hohe Schmelztemperatur und sind somit ideal zum Nachweis kurzer oder sehr ähnlicher Zielsequenzen.
Auch AT-reiche Oligonucleotide, die geringe Schmelztemperaturen aufweisen, können durch den Einbau von LNA-Nucleotiden ihre Tm erhöhen.
Tm Normalisierung
Für manche Anwendungen, bei denen die Bindung an viele unterschiedliche Targets gleichzeitig stattfindet (z.B. Microarrays), ist ein enger Tm-Bereich vorteilhaft. Unterschiede im GC-Gehalt der Zielsequenzen (z.B. miRNA) erschweren jedoch häufig die Detektion und Analyse mit traditionellen Nachweismethoden.
Der gezielte Einbau von LNA-Nucleotiden kann die Tm aller beteiligten Sonden jedoch soweit anpassen
(Tm Normalisierung), dass ein spezifisches Binden unterschiedlicher Targets bei gleichen Reaktionsbedingungen möglich ist.
Weitere Vorteile in der Anwendung
Vergleichende Untersuchungen zeigten, dass LNA/DNA-Hybride im Gegensatz zu PMOs (phosphorodiamidate morpholino oligo, neutral geladenes Rückgrat) aufgrund ihres negativ geladenen Rückgrats einfacher in Zellen gelangen und hierdurch auch intrazelluläre Anwendungen erlauben, z.B. Inhibierung der Translation mithilfe LNA-Antisense-Oligonucleotiden (siLNA).5
Ein wichtiger praktischer Vorteil im Umgang mit LNA-Oligos ist zudem, dass diese in Wasser löslich sind, und somit deren Handhabung enorm vereinfachen.4
Wo finden LNA-Oligonucleotide Anwendung?
Aufgrund ihrer vielfältigen Eigenschaften finden LNA-Oligonucleotide in weiten Bereichen der Molekularbiologie Anwendung:
- PCR / qPCR Anwendungen
- Dual-labelled Probes, Molecular Beacons, Scorpions, auch Multiplexing möglich
- Allel-spezifische PCR - in situ Hybridisierung (LNA-FISH)
- Microarrays
- Detektion von microRNA Sequenzen
- verbesserte Unterscheidung verschiedener microRNA Familien (unabhängig vom GC-Gehalt) - Nachweis einzelner Nucleotid-Mismatche (SNP Detektion)
- Genexpressionsanalyse
- Antisense Oligonucleotide (Gene silencing mit siLNA)
- Aptamere
- Pathogen-Detektion
- Methylierungsanalysen
- Mutagenese-Untersuchungen
Literatur:
1. LNA (Locked Nucleic Acids): Synthesis of the adenine, cytosine, guanine, 5-methylcytosine, thymine and uracil bicyclonucleoside monomers, oligomerisation, and unprecedented nucleic acid recognition. Koshkin AA, Singh SK, Nielsen P, Rajwanshi VK, Kumar R, Meldgaard M, Olsen CE, Wengel J; Biochemistry (2006), 45 (23), S. 7447–7455; doi:10.1021/bi060307w.
2. Synthesis of 2′-O,4′-C-methyleneuridine and -cytidine. Novel bicyclic nucleosides having a fixed C3, -endo sugar puckering. Obika S, Nanbu D, Hari Y, Morio KI, In Y, Ishida T, Imanishi T; Tetrahedron Letters (1997), 38 (50), S. 8735–8738; doi:10.1016/S0040-4039(97)10322-7.
3. Structures, dynamics, and stabilities of fully modified locked nucleic acid (β-D-LNA and α-L-LNA) duplexes in comparison to pure DNA and RNA duplexes. Suresh G, Priyakumar UD; J Phys Chem B. (2013), 117(18):5556-64. doi: 10.1021/jp4016068.
4. FISHing with locked nucleic acids (LNA): evaluation of different LNA/DNA mixmers. Silahtaroglua AN, Tommerupa N, Vissing H; Molecular and Cellular Probes 17 (2003) 165–169.
5. LNA/DNA mixmer-based antisense oligonucleotides correct alternative splicing of the SMN2 gene and restore SMN protein expression in type 1 SMA fibroblasts. Touznik A, Maruyama R, Hosoki K, Echigoya Y, Yokota T; Sci Rep. (2017); 7: 3672, doi: 10.1038/s41598-017-03850-2.
6. Biological Activity and Biotechnological Aspects of Locked Nucleid Acids. Lundin KE, Højland T, Hansen BR, Persson R, Bramsen JB, Kjems J, Koch T, Wengel J, Smith CI; Adv Genet. (2013), 82:47-107. doi: 10.1016/B978-0-12-407676-1.00002-0.
7. Design of antisense oligonucleotides stabilized by locked nucleic acids. Kurreck J, Wyszko E, Gillen C, Erdmann VA; Nucleic Acids Res. (2002); 30(9): 1911–1918.
8. Detection of mRNA in living cells by double-stranded locked nucleic acid probes. Riahi R, Dean Z, Wu TH, Teitell MA, Chiou PY, Zhang DD, Wong PK; Analyst. (2013), 138(17):4777-85. doi: 10.1039/c3an00722g.
9. LNA (Locked Nucleic Acid): High-Affinity Targeting of Complementary RNA and DNA. Vester B, Wengel J; Biochemistry (2004), 43 (42), pp 13233–13241, DOI: 10.1021/bi0485732.
10. Locked nucleic acid (LNA): fine-tuning the recognition of DNA and RNA. Braasch DA, Corey DR; Chemistry & Biology (2001), Vol. 8, pp 1-7.
11. Enhanced allele‐specific PCR discrimination in SNP genotyping using 3′ locked nucleic acid (LNA) primers. Latorra D, Campbell K, Wolter A, Hurley JM; Human Mutation (2003), Volume 22, pp 79-85.
12. Oligonucleotide-based systems: DNA, microRNAs, DNA/RNA aptamers. Jolly P, Estrela P, Ladomery M; Essays Biochem. (2016), 60(1): 27–35, doi: 10.1042/EBC20150004.
Designregeln
Allgemeine Regeln für das Design von Locked Nucleic Acid (LNA)
Im Gegensatz zu Standard DNA- oder RNA-Oligonucleotiden sind Oligonucleotide mit LNA-Basen aufgrund ihrer signifikant höheren Schmelztemperatur und der damit verbundenen höheren Sensitivität tendenziell eher kürzer.
Beim Design von Oligonucleotiden mit LNA-Basen gelten zunächst die allgemein gültigen Regeln für das Design von „normalen“ Oligonucleotiden:
- Der GC-Gehalt sollte zwischen 30-60% liegen.
- Bei Primer-Paaren sollten die Werte der Tm nahezu gleich sein.
Zusätzlich können die folgenden Punkte als grobe Richtlinien angesehen werden:
- Mehr als 4-5 hintereinander liegende LNA-Basen (außer bei sehr kurzen Oligos) sowie Stretche von 3 +G oder +C sollten vermieden werden.1
- Am 3´-Ende bzw. nahe des 3´-Terminus sollten nicht mehrere LNA-Basen positioniert werden.1
- LNA-Basen sollten nicht innerhalb palindromischer Sequenzen liegen, da in diesem Fall die Bindung innerhalb des Oligos stärker sein kann als an das eigentliche Target.2
- Der Einbau eines einzelnen LNA-Bausteins kann die Tm um 2-6°C bezüglich der Bindung an DNA und um 3-9,6°C bezüglich der Bindung an RNA erhöhen.5
- Werden mehrere LNA-Bausteine pro Oligonucleotid eingesetzt, verringert sich der Einfluss eines LNA-Monomers
auf die Tm. - Terminale LNA-Bausteine haben nur einen geringen Einfluss auf die Tm.3
- Die Tm wird nicht bzw. kaum beeinflusst, wenn das Oligonucleotid nur einen LNA-Baustein am 5´-Ende aufweist. 4
- Insgesamt sollten mehr als 15 LNA-Basen innerhalb eines Oligos vermieden werden, da dies eine Selbst-Hybridisierung fördert. Der LNA-Gehalt wird bei längeren Oligonucleotiden (>15 Basen) entsprechend reduziert.
Literatur:
Allgemein
1. Biological Activity and Biotechnological Aspects of Locked Nucleid Acids. Lundin KE, Højland T, Hansen BR, Persson R, Bramsen JB, Kjems J, Koch T, Wengel J, Smith CI; Adv Genet. (2013), 82:47-107. doi: 10.1016/B978-0-12-407676-1.00002-0.
2. Single nucleotide polymorphism genotyping using short, fluorescently labeled locked nucleic acid (LNA) probes and fluorescence polarization detection. Simeonov A, Nikiforov TT; Nucleic Acids Res. (2002), 30(17):e91.
3. Design of antisense oligonucleotides stabilized by locked nucleic acids. Kurreck J, Wyszko E, Gillen C, Erdmann VA. Nucleic Acids Res. 2002 May 1;30(9):1911-8.
4. Stopped-flow kinetics of locked nucleic acid (LNA)-oligonucleotide duplex formation: studies for LNA-DNA and DNA-DNA interactions. Christensen U, Jacobsen N, Rajwanshi VK, Wengel J, Koch T; Biochem J. (2001); 354(Pt 3):481-4.
5. Oligonucleotide-based systems: DNA, microRNAs, DNA/RNA aptamers. Jolly P, Estrela P, Ladomery M; Essays Biochem. (2016), 60(1):27-35. doi: 10.1042/EBC20150004.
6. Comparison of different real-time PCR chemistries and their suitability for detection and quantification of genetically modified organisms. Buh Gasparic M, Cankar K, Zel J, Gruden K; BMC Biotechnol. (2008), 8:26. doi: 10.1186/1472-6750-8-26.
7. Novel locked nucleic acid (LNA)-based probe for the rapid identification of Chlamydia suis using real-time PCR. Lis P, Kumala A, Spalinski M, Rypula K; BMC Vet Res. (2014), 10: 225. doi: 10.1186/s12917-014-0225-4.
in situ Hybridisierung
8. Detection of mRNA in living cells by double-stranded locked nucleic acid probes. Riahi R, Dean Z, Wu TH, Teitell MA, Chiou PY, Zhang DD, Wong PK; Analyst. (2013), 138(17):4777-85. doi: 10.1039/c3an00722g.
9. In situ detection of miRNAs in animal embryos using LNA-modified oligonucleotide probes. Kloosterman WP, Wienholds E, de Bruijn E, Kauppinen S, Plasterk RH; Nat Methods. (2006), 3(1):27-9.
10. A Sensitive Alternative for MicroRNA In Situ Hybridizations Using Probes of 2′-O-Methyl RNA + LNA. Søe MJ, Møller T, Dufva M, Holmstrøm K; J Histochem Cytochem. (2011), 59(7): 661–672, doi: 10.1369/0022155411409411.
Antisense-Oligos
11. Potent and nontoxic antisense oligonucleotides containing locked nucleic acids. Wahlestedt C, Salmi P, Good L, Kela J, Johnsson T, Hökfelt T, Broberger C, Porreca F, Lai J, Ren K, Ossipov M, Koshkin A, Jakobsen N, Skouv J, Oerum H, Jacobsen MH, Wengel J; Proc Natl Acad Sci U S A. (2000), 97(10):5633-8.
SNP Microarray Analysen
12. Design of LNA probes that improve mismatch discrimination. You Y, Moreira BG, Behlke MA, Owczarzy R; Nucleic Acids Res. (2006), 34(8):e60.
Molecular Beacons
13. Synthesis and investigation of deoxyribonucleic acid/locked nucleic acid chimeric molecular beacons. Yang CJ, Wang L, Wu Y, Kim Y, Medley CD, Lin H, Tan W; Nucleic Acids Res. (2007), 35(12): 4030–4041, doi: 10.1093/nar/gkm358.
Hydrolysesonden
14. A locked nucleic acid (LNA)-based real-time PCR assay for the rapid detection of multiple bacterial antibiotic resistance genes directly from positive blood culture. Zhu L, Shen D, Zhou Q, Li Z, Fang X, Li QZ; PLoS One. (2015), 10(3):e0120464. doi: 10.1371/journal.pone.0120464.
Bestellanleitung
Bestellanleitung für Locked Nucleic Acid Probes
|


