
Reinigung
Im Folgenden werden die verschiedenen Reinigungsarten kurz beleuchtet, die biomers.net anbietet. Nicht bei jedem Oligo kann die Reinigung gewählt werden (z. B. werden Farbstoff-modifizierte Oligos immer über HPLC gereinigt), bei unmodifizierten Oligos kann es dagegen sinnvoll sein, in Abhängigkeit von der Länge und von der Verwendung unterschiedliche Reinigungsarten zu wählen.
Kartusche
RP (reverse phase) Kartusche
Standardreinigung
Bei der Oligonucleotidsynthese wird die terminale, stark hydrophobe Dimethoxytrityl-Schutzgruppe am 5’-Ende des Oligos (DMT on) zunächst belassen. Sie dient als Unterscheidungskriterium zwischen den so genannten Fehlsequenzen (kürzere Oligoketten) und der eigentlichen Zielsequenz (Oligo mit voller Länge).
Nach Träger- und Schutzgruppenabspaltung wird die wässrige Lösung des Oligonucleotid-Rohprodukts auf das Kartuschenmaterial gegeben. Die Oligonucleotidketten mit voller Länge (DMT on) werden stark an das Kartuschenmaterial adsorbiert (=zurückgehalten, deshalb auch 'reverse phase', weil das gewünschte Produkt erst am Schluss von der Säule eluiert wird), die kürzeren Fehlsequenzen (DMT off) dagegen nicht. Durch Spülschritte können diese, wie auch eventuelle Salze etc., sehr leicht weggewaschen werden. Mit einer Säurebehandlung wird nun die DMT-Gruppe vom 5’-Ende des Zieloligonucleotids abgespalten, wonach dieses mit Wasser/Acetonitril vom Kartuschenmaterial eluiert werden kann. Es resultiert ein gereinigtes, salzfreies Oligonucleotid.
Bei kurzen, unmodifizierten (bis etwa 30 Basen) Oligonucleotiden lassen sich durch Kartuschenreinigung sehr gute Ergebnisse erzielen, nahezu vergleichbar einer HPLC-Reinigung. Alle kurzen Oligonucleotide (bis etwa 40 Basen) werden nach der Reinigung einer Endkontrolle mit Maldi Massenspektrometrie unterzogen.
RP-HPLC
HPLC-Reinigung von Oligonucleotiden
Auch bei der HPLC wird über eine 'reverse phase'-Säule gereinigt. Da sich das Verhältnis der Hydrophobizität von vollständigen (DMT on, siehe auch Reinigung über Kartusche) und unvollständigen (DMT off) Sequenzen mit zunehmender Länge der Oligonucleotide zu Ungunsten der Vollständigen verschiebt, ist mit der HPLC eine längere Trennstrecke gegeben, um diesem Effekt entgegenzuwirken. Für längere Oligonucleotide (>30 Basen) ist eine Aufreinigung durch 'reverse phase' HPLC empfehlenswert. Je nach Länge der Oligonucleotide kann durch geeignete Wahl der Trennsäulen (Länge, Material) und Elutionsgradienten eine optimale Separation, auch hier über die DMT-Schutzgruppe, von Produkt und Verunreinigungen erreicht werden.
Der Reinigungsverlauf wird UV-spektroskopisch (260 nm) verfolgt und aufgezeichnet, so dass der Produktpeak nicht „von Grundlinie zu Grundlinie“ gesammelt wird, sondern „auf Qualität geschnitten“, d.h. eventuelle Schultern werden nicht gesammelt.
Für endmodifizierte Oligonucleotide ist eine HPLC-Reinigung ebenfalls die Methode der Wahl. Die Reinigung von Farbstoff-modifizierten Oligonucleotiden wird bei verschiedenen Wellenlängen kontrolliert, üblicherweise bei 260 nm (Oligo) und im Absorptionsmaximum des jeweiligen Farbstoffs.
Mehrfach markierte Oligonucleotide (Dual-Labelled Probes, Molecular Beacons) werden einem mehrstufigen Reinigungsprozess unterzogen.
IEX-HPLC
IEX-HPLC
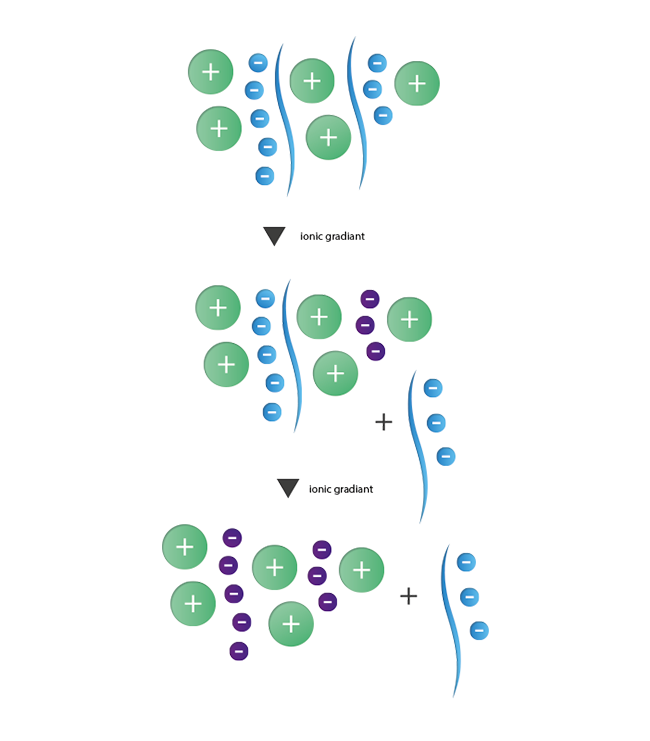
Bei der Ionentauscherchromatographie beruht das Trennprinzip auf der Wechselwirkung zwischen unterschiedlich geladenen Teilchen. Mit ihr ist es möglich, Spezies mit einer unterschiedlichen Anzahl von Ladungen voneinander zu trennen. Zur Reinigung von Oligonucleotiden bietet sich daher ein Anionentausch an, besteht deren Rückgrat doch aus negativ geladenen Phosphatgruppen. Als Matrix für die stationäre Phase wird ein Harz mit positiv geladenen Gruppen verwendet. Diese treten dann in Wechselwirkung mit den negativen Ladungen der Oligonucleotide. Die Separation von Strängen mit der Länge N von Abbruchsequenzen mit der Länge N-1, N-2, usw. erfolgt über einen Gradienten zunehmender Ionenstärke. Dabei werden die niedriger geladenen Abbruchsequenzen zuerst eluiert, während die Produktsequenzen mit der vollen Länge noch zurückgehalten werden und als letztes eluieren. Die Bedeutung der Ionentauscherchromatographie als alternative bzw. zusätzliche Reinigungsmethode für Oligonucleotide hat in den letzten Jahren stetig zugenommen.
 |
PAGE
Polyacrylamid Gel-Elektrophorese (PAGE)
Während bei den bisher beschriebenen Methoden anhand des Unterscheidungskriteriums 'DMT-Gruppe' getrennt wird, findet bei der Polyacrylamid Gel-Elektrophorese (PAGE) eine Separation nach Ladung bzw. Molekulargewicht statt.
Die vollständig entschützten Oligonucleotide werden je nach Länge über unterschiedlich stark vernetzte denaturierende Polyacrylamidgele getrennt. Dabei kann im Idealfall eine Auflösung von einem Nucleotid erreicht werden.
Nach der Elektrophorese werden die einzelnen Banden durch „UV-Shadowing“ sichtbar gemacht und die gewünschte Bande aus dem Gel ausgeschnitten. Mittels Wasser bzw. Pufferlösung wird das Oligonucleotid aus dem Gelmaterial eluiert, entsalzt und quantifiziert.
Insbesondere für sehr lange Oligonucleotide, die in Klonierungs- und Mutagenese-Experimenten eingesetzt werden, ist die PAGE-Reinigung die Methode der Wahl.

