
Enzymatische Kopplungen über Oligonucleotide mit
Protein-Tag-spezifischen Liganden
Einleitung
Enzymatische Kopplungen über Oligonucleotide
Seit Jahren hat die Erforschung der Struktur und Funktion der Proteine einen besonders hohen Stellenwert in der Forschung. Mit der Etablierung rekombinanter DNA-Systeme können Proteine in artfremden Organismen leicht exprimiert und isoliert werden. Hierbei haben sich bestimmte Protein-Tags als vielversprechend erwiesen.
Am gesuchten Protein gebundene, sogenannte Epitop-Affinitäts-Tags ermöglichen eine gezielte Aufreinigung rekombinanter Proteine aus dem Zellextrakt. Hierzu gehören beispielsweise der His-Tag (Poly-Histidin), GST-Tag (Glutathion S-Transferase), MBP-Tag (Maltose Binding Protein), CBP-Tag (Calmodulin Binding Protein) oder der FLAG-Tag, mit dessen Hilfe über eine Affinitätschromatografie eine Erhöhung der Proteinreinheit und -ausbeute erzielt werden kann. 1
Protein-Tags der neueren Generation, wie der SNAP- oder CLIP-Tag sowie der HaloTag®, zeichnen sich hingegen durch ein weitaus breiteres Einsatzspektrum aus und finden in verschiedenen Bereichen Anwendung:
- Detektion
- Lokalisierung
- Protein-Protein-Interaktionen
- Labelling, Multiplex Labelling (z.B. SNAP-/CLIP-Tag)
- enzymatische Verknüpfung verschiedener Proteine oder anderer Biomoleküle
Bei biomers.net erhalten Sie Oligonucleotid-Liganden für eine Vielzahl an Protein-Tags (HaloTag®, SNAP-Tag, Clip-Tag, etc.).
Zur weiteren Analyse der Fusionsproteine werden die Oligonucleotide hierbei mit Tag-spezifischen Liganden markiert.
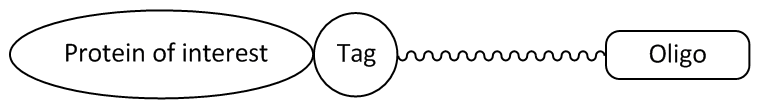
Abbildung 1: Oligonucleotid-Ligand mit einer Tag-spezifischen Linkerstruktur, die vom Fusionsprotein kovalent gebunden wird.
Protein-Tags der neueren Generation sind im Allgemeinen kleinere Proteine, die mittels Translation an ein Targetprotein angefügt werden können.
Die grundsätzliche Funktionsweise der Protein-Tags wie dem HaloTag® oder dem SNAP- bzw. CLIP-Tag, beruht auf der Erkennung eines bestimmten Substrates oder Liganden (z.B. Fluoreszenz-Konjugat aus Oligonucleotid und Fluoreszenzfarbstoff). Dieses kann hochspezifisch vom Tag erkannt und kovalent gebunden werden, woraufhin weitere Untersuchungen des Fusionsproteins ermöglicht werden. 2
Durch Austausch des Oligonucleotid-Liganden kann die Funktion sowie die Eigenschaften des Fusionsproteins beeinflusst werden (z.B. Membrangängigkeit, Fluoreszenzmarkierung, Oberflächenbindung, etc.).
Literatur:
1. Overview of Affinty Tags for Protein Purification. Kimple ME, Brill AL, Pasker RL; Curr Protoc Protein Sci. (2015); 73,: Unit-9-9.
2. Snap-, CLIP- and Halo-Tag Labelling of Budding Yeast Cells. Stagge F, Mitronova GY, Belov VN, Wurm CA, Jakobs S; Plos One (2013); Volume 8, Issue 10, e78745.
3. Motor reattachment kinetics play a dominant role in multimotor-driven cargo transport. Feng Q, Mickolajczyk KJ, Chen G-Y, Hancock WO; CC-BY-NC-ND 4.0 International license.
SNAP-/CLIP-Tag
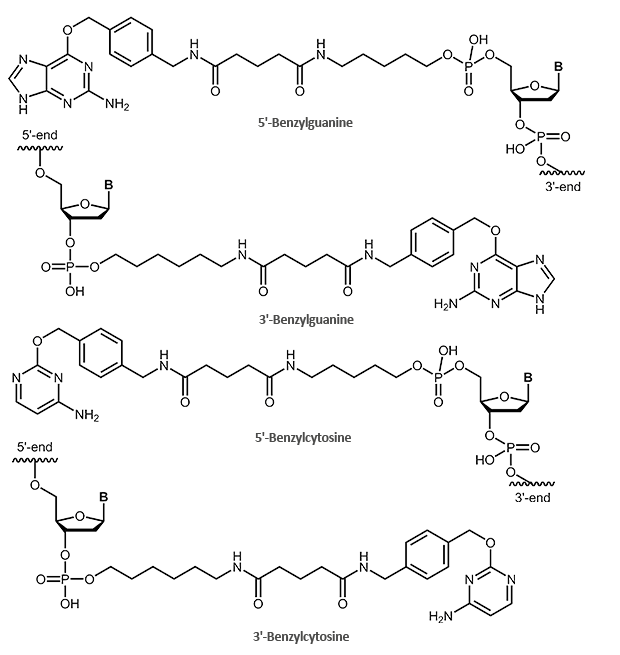
SNAP- und CLIP-Tag Protein-Liganden – Benzylguanin- und Benzylcytosin-markierte Oligonucleotide
Es gibt eine Vielzahl an möglichen Tools, die ein selektives Proteinlabelling in lebenden Zellen erlauben. Hierbei haben sich neben dem Halo-Tag, der SNAP- und der CLIP-Tag hervorgehoben. Beide Tags erlauben die Visualisierung komplexer, zellulärer Prozesse (z.B. Protein-Protein-Interaktionen, Proteinlokalisierung, Proteinfunktionsanalysen).
Auch hier wird mittels Vektorexpressionssystem der Protein-Tag an das gesuchte „protein of interest“ fusioniert. Der Tag kann dabei N- oder C-terminal an das Targetprotein gebunden werden.
Der SNAP-Tag selbst ist ein kleines Protein und leitet sich von dem 20 kDa großen, humanen DNA Reparaturprotein O6-Alkylguanin-DNA Alkyltransferase (AGT) ab. Als Substrat dient das O6-Benzylguanin (BG) Derivat.
Hierbei wird die Benzylgruppe des BG-Derivats auf das Cystein im aktiven Zentrum des Protein-Tags transferiert, sodass Tag und BG-Derivat kovalent miteinander verbunden werden. Die Guanin-Gruppe wird dabei abgespalten.
Eine modifizierte Form des SNAP-Tags ist der CLIP-Tag. Dieser basiert ebenfalls auf dem Enzym AGT, reagiert jedoch spezifisch mit O2-Benzylcytosin (BC) Derivaten.
Aufgrund der orthogonalen Substratspezifität und der fehlenden Kreuzreaktivität der beiden Tags untereinander, ist ein sogenanntes Multicolour Labelling möglich. Hierbei können SNAP- und CLIP-Tag-Fusionsproteine gleichzeitig für das Markieren von mehreren Proteinen in einer Zelle genutzt werden.
Oligonucleotide mit SNAP- und CLIP-Tag-Liganden
Mit biomers.net können Sie Ihre Oligos am 5´- oder 3´-Ende mit einem passenden Protein-Tag-Liganden markieren. Hierfür wird das Oligo entweder mit einem Benzylguanin (SNAP-Tag) oder einem Benzylcytosin (CLIP-Tag) markiert. Das Oligo selbst kann dabei auch weiter modifiziert werden (Fluoreszenzfarbstoffe, Biotin).
Literatur:
1. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Gautier A, Juillerat A, Heinis C, Reis Corrêa I, Kindermann M, Beaufils F, Johnsson K; Chemistry and Biology 15 (2008), 128-136, DOI 10.1016.
2. Simultaneous Protein Tagging in Two Colors. Schultz C, Köhn M; Chemistry & Biology (2008), 91-2. doi: 10.1016/j.chembiol.2008.02.004.
Sortase/Triglycin
Sortase-vermittelte Verknüpfungsreaktion über Triglycin-modifizierte Oligos
Allgemein handelt es sich bei Sortasen um bakterielle, Membran-assoziierte Enzyme aus der Klasse der Transpeptidasen. Ihre natürliche Funktion umfasst neben dem Strukturaufbau bakterieller Pili, auch die kovalente Verknüpfung sezernierter Proteine aus dem Cytoplasma in die Peptidoglykanschicht gram-positiver Bakterien. 1,2
Beim Aufbau der Zelloberfläche werden neben strukturgebenden Proteinen auch Virulenzfaktoren in die Zellwand integriert, die für die Pathogenität der Mikroorganismen verantwortlich sind. Die Inaktivierung der beteiligten Sortase kann die Virulenz der Bakterien beeinflussen oder sogar inhibieren.3,4
Seit einigen Jahren findet die Transpeptidasereaktion der Sortasen auch in vitro Anwendung:
- kovalente Verknüpfung verschiedener Proteine oder anderer Biomoleküle
- Analyse der Bindungseigenschaften eines Proteins z.B. mittels Farbstoff-markierter Antikörper oder Liganden
- PEGylierung von Molekülen in der Therapeutik: Kopplung von Polyethylenglycol (PEG) an ein Biomolekül verhindert dessen intrazellulären Abbau und erhöht somit die pharmakodynamischen Eigenschaften 5
- Ringschluss von Proteinen und somit Inhibierung der Degradation 6
Die Verbindung beruht auf einem bestimmten terminalen Erkennungsmotiv am Targetprotein – das LPXTG-Motiv (Leucin-Prolin-Xaa-Threonin-Glycin). X steht hierbei für eine beliebige Aminosäure.
Sortasen sind in der Lage die LPXTG-Sequenz am Peptid zu spalten, um es dann im Folgenden mit dem gewünschten Liganden (Oligonucleotid, Peptid, Protein, etc.) zu verbinden.
In einem ersten Schritt entsteht ein Intermediärprodukt aus Sortase und Targetprotein. Hierfür bindet der Thiolrest des Cysteins im aktiven Zentrum der Sortase das Threonin innerhalb der Erkennungssequenz, wobei der C-terminale Glycinrest des Proteins abgespalten wird. Im nächsten Schritt bindet die Carboxylgruppe des Threonins die Aminogruppe des freien terminalen Triglycins, sodass Targetprotein und Biomolekül kovalent verbunden werden.7
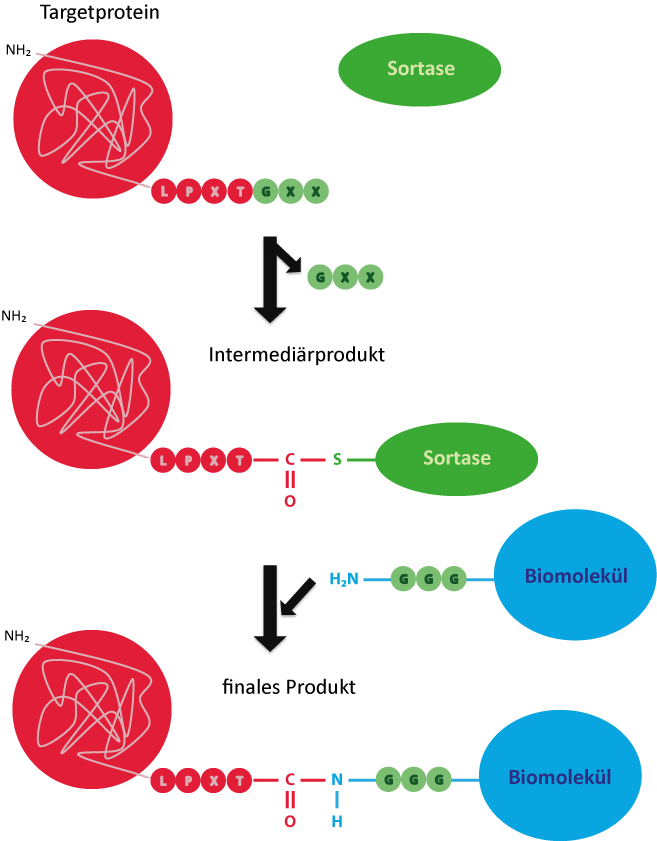
Abbildung 3: Reaktionsmechanismus der Sortase zur kovalenten Verknüpfung eines Targetproteins mit einem Triglycin-markierten Biomolekül.
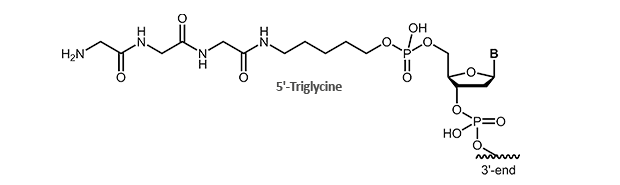
Für diese Anwendung bieten wir Ihnen Triglycin-modifizierte Oligonucleotide an. Die Modifikation ist für das 5´-Ende des Oligonucleotids verfügbar.
Bei Fragen können Sie uns gerne jederzeit kontaktieren!
Literatur:
1. Sortase Transpeptidases: Insights into Mechanism, Substrate Specificity and Inhibition. Clancy KW, Melvin JA, McCafferty DG; Biopolymers (2010), 94(4): 385–396. doi: 10.1002/bip.21472.
2. Sortase enzymes in Gram-positive bacteria. Spirig T, Weiner EM, Clubb RT; Mol Microbiol. (2011), 82(5):1044-59. doi: 10.1111.
3. Sortase A: an ideal target for anti-virulence drug development. Cascioferro S, Totsika M, Schillaci D; Microb Pathog. (2014), 77:105-12. doi: 10.1016.
4. Inhibition of sortase-mediated Staphylococcus aureus adhesion to fibronectin via fibronectin-binding protein by sortase inhibitors. Oh KB, Oh MN, Kim JG, Shin DS, Shin J; Appl Microbiol Biotechnol. (2006), 70(1):102-6. Epub 2005 Jul 12.
5. Sortase-catalyzed transformations that improve the properties of cytokines. Popp MW, Dougan SK, Chuang T-Y, Spooner E, Ploegha HL; Proc Natl Acad Sci U S A (2011), 108(8): 3169–3174.
6. Sortase-mediated backbone cyclization of proteins and peptides. van 't Hof W, Hansenová Maňásková S, Veerman EC, Bolscher JG; Biol Chem. (2015), 396(4):283-93. doi: 10.1515/hsz-2014-0260.
7. Sortase-mediated protein ligation: a new method for protein engineering. Mao H, Hart SA, Schink A, Pollok BA; J Am Chem Soc. (2004), 126(9):2670-1.

